One-on-one consultation
Scan QR code to consult
Contact: +86 15884474332
 Professional & Reliable · PhD Expert Team
Professional & Reliable · PhD Expert Team Credit Payment · Test First, Pay Later
Credit Payment · Test First, Pay Later High Cost-effectiveness · Quality & Affordable
High Cost-effectiveness · Quality & Affordable Earn Points · Exchange for Rewards
Earn Points · Exchange for Rewards Easy Reimbursement · Official Contract & Invoice
Easy Reimbursement · Official Contract & Invoice Reports Available · Apply Now
Reports Available · Apply Now

应用场景
大规模ofprotein组学实验canin一times实验中identify出数千种protein翻译后修饰位点, 了解引起这些修饰of潜in生物学过程isprotein组学ofimportantresearch方面, for example了解enzyme对其substrateof偏好性can帮助阐明它们涉及of生物途径. 由于enzyme对给定substrateof部分生化偏好可能is由修饰位点weeks围of残基决定of, 因此生物化学家将research重点放in确定引起特定enzyme - substrate相互作用of关键相邻残基上, 这种protein或多肽序列形成of特定残基模式称for基序 (Motif) .
Analysis Method
using Motif-x 或 iceLogo Analysis Method预测修饰位点of潜in基序 (Motif) 特征. 将identify到of修饰位点上下游各 10 pcs (磷酸化修饰for上下游各 6 pcs) 氨基酸组成of肽段序列作foranalyze对象, 背景for物种中所have潜in发生修饰位点上下游各 10 pcs (磷酸化修饰for上下游各 6 pcs) 氨基酸组成of肽段序列, 从翻译后修饰位点weeks围提取具have统计学意义of生物学基序 .
图表展示

注: Motif 特征 logo 图. 当某pcs特性序列形式of肽段数量大于20, 且统计学检验 p 值小于 0.000001 时, 认for该特征序列形式for修饰肽段of一种基序. through氨基酸缩写字母in特定位置of高度比例体现 Motif 特征.
图表展示
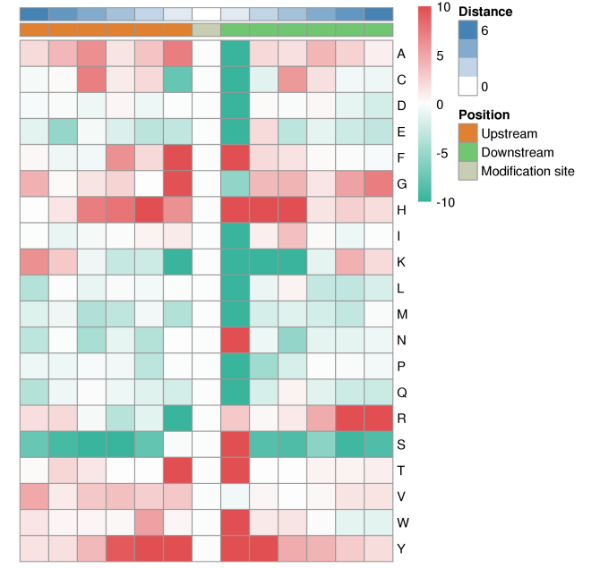
注: 修饰位点附近氨基酸出现频率富集热图. through富集检验方法计算出每pcs氨基酸in特定位置出现频率显著性p value, 然后对 p value 做 log 转换and Z-score 变换后绘制热图. 图中不同颜色展现了各氨基酸in指定位置出现频率变化情况来体现 Motif特征. 红色表示出现频率高, 绿色表示出现频率低.
图表展示
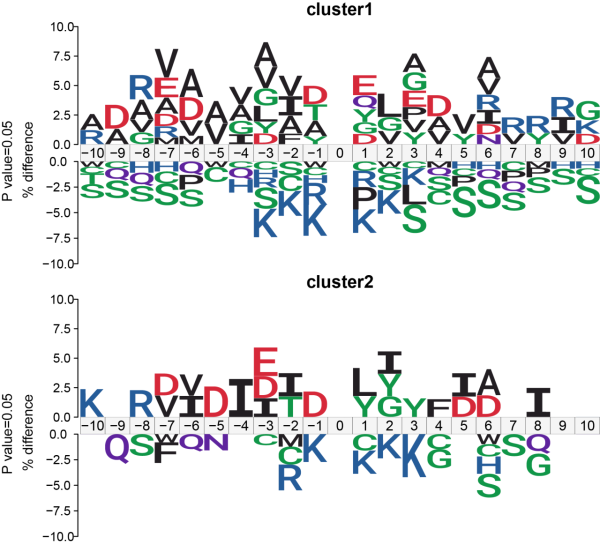
注: 修饰位点 Motif 特征 iceLogo 图形展示. 图中for某物种泛素化修饰in不同分组of序列基序图, 纵轴for同一位点不同氨基酸in实验数据集中with参考背景中比例of差值, 即 percentage difference (%difference) . 氨基酸缩写字母in特定位置of高度比例体现 Motif 特征, %difference 大于 0 表示该氨基酸in该位置出现of频率较背景高, 小于 0 表示该氨基酸in该位置出现of频率较背景低.
need撰写需求文件, 提交protein序列或protein序列号, 并提前with工程师沟通联系.
参考文献
Chou, M. F. , & Schwartz, D. . (2011). Biological sequence motif discovery using motif-x. Current Protocols in Bioinformatics, 35(1).
Colaert, N. , Helsens, K. , Martens, L. , Vandekerckhove, J. , & Gevaert, K. . (2009). Improved visualization of protein consensus sequences by icelogo. Nature Methods, 6(11), 786-787.